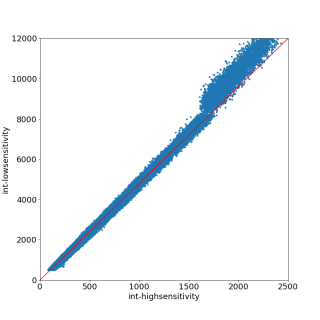
リガクのIPは8192カウントで高感度と低感度の切り替えがあります。それらの繋ぎをスムーズにするために、HV/ADVで25keV-2mAで1秒と10秒で測定したところ、direct 5200/52000countでちょうど10倍になっていたので、HV2の調整は行っていません。チェックのために無機物のファインパウダーを10度の振動写真で3時間と18時間測定し、同じ1pixcelにある強度同士の2Dプロットが上図。8192カウントで段差があります。 HV/ADVでは、読み込み時のフェーディングが考慮されていないためと思われます。シチジンのR1が下がっているため、とりあえずこのままとしました。以下に、飛びの有無をチェックするためのpythonのコードも添付します。ちなみに、この現象は放射光でも同様ですので、きちんと解析したい方は、Zrの吸収端等を用いて必ずチェックする必要があります。特に粉末回折では重大な影響があるので、1)厳密にHVを調整する、2)影響がないカウント以下で重ね取りする(8000カウント以下で撮ればよいとは限りません)、3)切り替え点前後で傾きのみの影響であれば、プログラムで補正する、などの方法をとります。
# -*- coding: utf-8 -*-
#RIGAKU imaging plate RAPID linearity check
#imaging plateは、1ピクセルあたり約8000-16000カウントでフォトマルの切り替えがありますが、
#定期的に調整しないとそのつなぎが飛んでいたりすることがあり、解析に悪影響を及ぼします。
#元になるデータは、CeO2などの粉末データを1ピクセルあたり8000カウント以下になる時間で
#10度振り振動写真を撮影(high_file)、もう1枚を1.5万から3万カウントになる時間で撮影し(low_file)、
#両者をRIGAKU-Displayから画像データをtxt形式に変換したものを使います
#共同利用者以外への利用責任はなく質問受付はしません。D2matEのプロジェクト支援を受けて行われています。
import numpy as np
import matplotlib.pyplot as plt
#file names
high_file = ‘****_103.txt’ # short time scan
low_file = ‘****_104.txt’ # long time scan
#data read
with open(high_file) as f:
high_list = f.read().split()
high_1d = np.array(high_list[2::3], dtype=’int16′)
with open(low_file) as f:
low_list = f.read().split()
low_1d = np.array(low_list[2::3], dtype=’int16′)
#conbine low- and high-sensitivity data
df = np.vstack([low_1d,high_1d]).T
stack_data = df[np.any(df > 500, axis=1)]
xscale = 2500
yscale = 12000
x = np.linspace(0, xscale, 10)
y = yscale/xscale*x
plt.figure(figsize=(10,10))
plt.xlim(0, xscale)
plt.ylim(0, yscale)
plt.rcParams[“font.size”] = 18
plt.xlabel(‘int-highsensitivity’)
plt.ylabel(‘int-lowsensitivity’)
plt.scatter(stack_data[:,1], stack_data[:,0], marker=”.”)
plt.plot(x, y, color = “red”)
plt.savefig(“graph.png”)
#np.savetxt(‘stack_data.csv’, stack_data, delimiter=”,”, fmt=’%d’)